了解其结构和功能对于设计针对特定目标的抗体至关重要。开发有助于预测这些结构的深度学习模型的研究是一个引人入胜的、不断发展的话题。深度学习是基于堆叠人工神经网络层的机器学习的一个子集,其中处理用于从仿射变换和非线性激活函数数据中提取越来越高级的特征。1
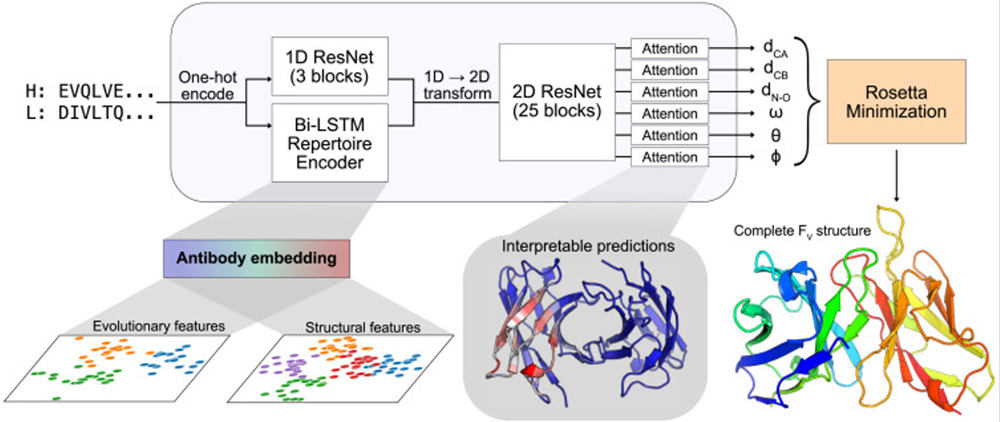
DeepH3 是一种深度学习模型,可预测抗体重链和轻链序列的残基间距离和取向。这些结果产生分布,然后将其转换为几何表示,并用于区分诱饵结构并预测新的抗体高变互补决定区 (CDR) H3 从头环。2
这些 CDR H3 环是抗原结合所必需的,即使序列相似性很高,它们也具有很高的构象多样性。3因此,CDR-H3 建模的约束比六个 CDR 中的其他五个(H1、H2、L1、L2 和 L3)少得多,这意味着识别高质量的 H3 模板可能是一项更艰巨的工作,而 DeepH3 模型非常有价值。
抗体结构深度学习方法的另一个例子是 DeepAb,它根据序列预测准确的抗体 FV 结构。Ruffolo等人有两个步骤。s (2022) 过程,第一个使用深度残差卷积网络来预测 FV 结构。其次,他们使用基于罗塞塔的快速协议从网络的预测中实现结构。该方法还可用于对单域 (VHH) 抗体进行建模。4
在Biointron,我们致力于加速您的抗体发现、优化和生产需求。我们的专家团队可以提供定制的解决方案,以满足您的特定研究需求。联系我们,详细了解我们的服务以及我们如何帮助您加速研究和药物开发项目。
- Gao,W.,Mahajan,SP,Sulam,J.和Gray,JJ(2020)。蛋白质结构建模和设计中的深度学习。模式,1(9),100142。https://doi.org/10.1016/J.PATTER.2020.100142
- Ruffolo,JA,Guerra,C.,Mahajan,SP,Sulam,J.和Gray,JJ(2020)。来自深度学习的几何势能改进了对 CDR H3 环路结构的预测。生物信息学, 36(Supplement_1), i268–i275.https://doi.org/10.1093/bioinformatics/btaa457
- Kim, J., McFee, M., Fang, Q., Abdin, O., & Kim, PM(2023 年)。基于计算和人工智能的抗体开发方法。药理学趋势,44(3),175-189。https://doi.org/10.1016/J.TIPS.2022.12.005
- Ruffolo,JA,Sulam,J.和Gray,JJ(2022)。使用可解释的深度学习进行抗体结构预测。模式,3(2),100406。https://doi.org/10.1016/J.PATTER.2021.100406