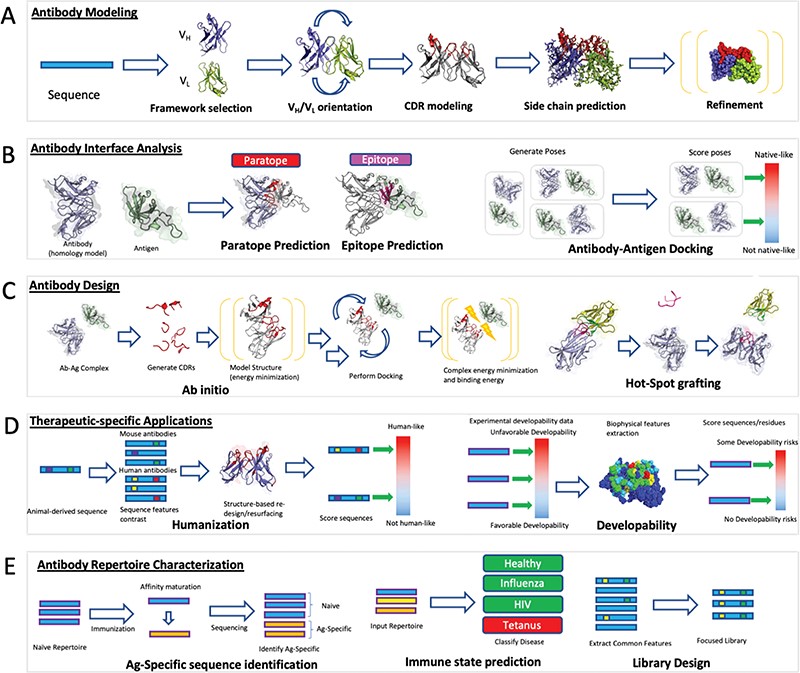
随着计算方法和人工智能(AI)的进步,治疗性抗体的开发得到了显着加强。这些技术简化了抗体发现过程,提高了识别和优化具有高特异性和有效性的抗体的能力。
人工智能在抗体设计中的作用
人工智能和机器学习现在是药物发现管道的核心参与者。它们有助于鉴定和优化新型候选抗体,具有以下几个关键优势:
- 生成式人工智能:通过高精度预测蛋白质结构,模型首次用于从头开始设计抗体。这为创造具有增强结合特性和治疗潜力的全新抗体支架提供了潜力。1生成式 AI 模型的一个例子是 DeepMind 开发的 AlphaFold 2。该程序在 2022 年 CASP(蛋白质结构预测的关键评估)竞赛中实现了近乎原子级的蛋白质结构预测精度。
- 改进的计算机模拟:人工智能允许对抗体-抗原相互作用进行稳健的计算机模拟。这有助于预测结合亲和力和虚拟筛选庞大的抗体库,从而大大减少了对费力的湿实验室实验的需求。
这些进步有望显著增强抗体的结合特性和治疗潜力。
关键计算方法
- 基于结构的设计:结构生物学的进步,特别是像AlphaFold这样的工具,已经彻底改变了抗体设计。这些工具可预测抗体的三维结构,使研究人员能够识别靶抗原上的关键结合位点,优化抗体序列以增强其对靶标的亲和力和特异性,并设计抗体以最大限度地减少脱靶结合和潜在的副作用。
- 机器学习模型:生物技术初创公司和制药公司现在正在利用大型数据集来预测潜在抗体的有效性和安全性。这些模型分析各种数据模式,包括遗传、蛋白质组学和临床数据,以确定有前途的治疗靶点并优化抗体序列。这种方法显著提高了抗体发现过程的效率。2
高通量筛选
计算方法改进了高通量筛选过程,可以快速评估大型候选抗体库。与自动化和机器人的集成还可以大大加快筛选过程,对样品进行一致的处理和处理,减少体力劳动并提高筛选通量。还可以设计计算管道来实时分析筛选数据。这使得研究人员能够根据新出现的数据调整筛选参数或确定特定候选药物的优先级,从而进一步优化筛选过程。
应用
最近的研究强调了这些计算方法的实际应用。例如,人工智能模型已被用于协助设计针对 COVID-19 的药物,展示了它们在应对新出现的传染病方面的潜力。例如,SARS-CoV-2突变的出现导致了影响疫苗有效性的新毒株,因此Thadani等人开发了EVEscape,这是一种深度学习生成模型,使用生物物理和结构信息在历史序列上进行训练。在预测 SARS-CoV-2 的大流行变异时,该模型表现出与高通量实验扫描相似的准确性。归根结底,预测可能的进一步突变以预测新出现的毒株是持续疫苗开发的非常有价值的工具。3
挑战与未来方向
尽管取得了重大进展,但抗体的计算设计仍存在一些挑战。确保人工智能模型在不同数据集和疾病背景下的通用性是一个主要障碍。此外,集成不同的数据类型和保持数据质量对于这些模型的成功至关重要。未来的研究应侧重于提高人工智能预测的可解释性,并开发强大的验证框架,以确保计算设计的抗体的临床相关性。
抗体的计算设计代表了药物发现的一种变革性方法,提供了前所未有的速度和精度。通过利用人工智能和机器学习的力量,研究人员可以开发更有效、更安全的治疗性抗体。计算科学家和生物学家之间的持续创新和合作对于充分实现这些技术在改善人类健康方面的潜力至关重要。
- Bennett, N., Watson, J., Ragotte, R., Borst, A., See, D., Weidle, C., Biswas, R., Shrock, E., Leung, P., Huang, B., Goreshnik, I., Ault, R., Carr, K., Singer, B., Criswell, C., Vafeados, D., Garcia Sanchez, M., Kim, H., Vázquez Torres, S., Chan, S., & Baker, D. 原子精确的单域抗体从头设计。生物Rxiv。(2024),https://www.biorxiv.org/content/early/2024/03/18/2024.03.14.585103
- Norman, RA, Ambrosetti, F., J Bonvin, MJ, Colwell, LJ, Kelm, S., Kumar, S., & Krawczyk, K. (2020)。治疗性抗体设计的计算方法:既定方法和新兴趋势。生物信息学简报,21(5),1549-1567。https://doi.org/10.1093/bib/bbz095
- Thadani, N. N., Gurev, S., Notin, P., Youssef, N., Rollins, N. J., Ritter, D., Sander, C., Gal, Y., & Marks, DS (2023).从大流行前的数据中学习以预测病毒逃逸。自然,622(7984),818-825。https://doi.org/10.1038/s41586-023-06617-0